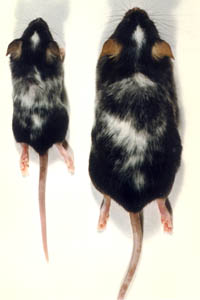
Aparición espontánea de una mutación Dominant Spotting en un ratón DBA/2J
Articulos Cientificos 24 April 2013Autor: Fernando Benavides
(1) Dirección actual: The University of Texas M.D. Anderson Cancer Canter, Science Park Research Division, 1808 Park Road 1C, Smithville, Texas, 78957, E-mail: fbenavid@mdanderson.org
*Trabajo realizado en:
División Medicina Experimental, Instituto de Investigaciones Hematológicas, Academia Nacional de Medicina, Pacheco de Melo 3081, 1425 Buenos Aires, Argentina
Introducción:
Las mutaciones identificadas hasta el momento en el ratón pueden ser de dos tipos: (i) Aquellas que aparecieron espontáneamente o después de un tratamiento mutagénico. Son miles, se traducen, en general, en un fenotipo patológico y podrían clasificarse como mutaciones tradicionales. (ii) Aquellas que resultan de una manipulación del genoma, como los ratones transgénicos, knock-out (KO) y knock-in (KI), también muy numerosas y en continuo aumento. Las mutaciones dirigidas, producidas por recombinación homóloga, conllevan normalmente la interrupción de una secuencia codificante y generan genes que no son traducidos a proteínas (alelos nulos). Por el contrario, los mecanismos que generan las mutaciones tradicionales son muy numerosos, de origen diverso, y aportan mucha información sobre la estructura y los mecanismos de regulación de los genes en los mamíferos (1). Muchas mutaciones generadas por agentes químicos son del tipo puntual (transiciones o transversiones de bases) y no implican más que una pérdida parcial de la actividad de la proteína (alelos hipomórficos). De esta manera, el estudio molecular de mutaciones tradicionales aporta casi siempre información original y transferible a la especie humana (2). Existen dos condiciones en los bioterios que favorecen el descubrimiento de mutaciones espontáneas: por un lado, la cría de roedores con sistemas de apareamiento consanguíneo y, por el otro, la observación diaria y atenta de los animales por parte de los técnicos e investigadores. Se considera que la frecuencia de mutaciones espontáneas es del orden de 5 x 10-6 por gameto y por generación para las mutaciones recesivas y de 2 x 10-7 para las dominantes. De todas formas, esta tasa debe ser considerada como un valor promedio ya que existen loci que mutan mucho más frecuentemente que otros. Por ejemplo, el locus Kit, referido en este artículo, presenta una tasa de mutación 10 veces más elevada que el promedio.
En el año 1991, en el bioterio de la entonces llamada División Medicina Experimental de la Academia Nacional de Medicina de Buenos Aires, un técnico descubrió –en su rutina diaria de cambio de jaulas- una hembra de la cepa consanguínea de ratones DBA/2 (D2) que presentaba manchas de pelaje blanco en la cabeza, dorso y abdomen. Se sospechó, por su fenotipo de manchas blancas que podría tratarse de la mutación Dominant White Spotting (W) (ahora KitW). Los resultados presentados aquí, permiten inferir que muy posiblemente se trataba de dicha mutación dominante, la cual apareció en forma espontánea.
Materiales y Métodos:
Para el presente estudio se desarrolló, a lo largo de seis años, una línea congénica con fondo C57BL/6 (B6) que denominaremos B6.D2-KitW. Estas líneas son producidas por la introducción de la región cromosómica que contiene un locus diferencial por retrocruzas repetidas de la cepa donante con una cepa receptora, a través de por lo menos 10 generaciones. De esta manera, en cada generación el nivel de heterocigosis se reduce un 50% y aumenta en la misma proporción el fondo genético de la cepa receptora (1). Para dicho propósito se realizaron 12 generaciones de retrocruzas (llegando hasta ratones N13), con selección del fenotipo de manchas blancas, utilizando la cepa D2 como dadora y la cepa B6 como receptora (Figura 1). El ADN de 60 ratones N13, 30 de fenotipo mutante (supuestamente KitW/+) y 30 de fenotipo salvaje (wild-type o genotipo +/+), fue extraído a partir de puntas de cola por métodos clásicos de fenol/cloroformo/isoamilalcohol. El ADN fue evaluado por PCR, utilizando “primers” específicos para el microsatélite D5Nds2 (obsequio del Dr. Jean-Louis Guénet, Institut Pasteur, París, Francia). Este marcador microsatélite es polimórfico entre las cepas parentales [alelo B6 = 166 pares de bases (pb) ; alelo D2 = 176 pb] y se ubica aproximadamente a 1 centiMorgan (cM) del locus Kit en el cromosoma 5 del ratón [Mouse Genome Database (MGD), Mouse Genome Informatics (MGI), The Jackson Laboratory, Bar Harbor, ME (URL:http://www.informatics.jax.org). November 2007]. Los microsatélites de ADN son repeticiones en tandem de 1-5 nucleótidos, en particular (CA)n y (GA)n. A estas secuencias se las conoce también como SSLP (“Simple Sequence Length Polymorfisms”) (1, 3, 4).
Las amplificaciones fueron hechas a partir de 100 ng de ADN en un volumen final de 25 µl, con una concentración final de 1,5 mM Cl2Mg, 200 µM de cada dNTP, 180 µM de cada primer y 0.5 U de Taq polimerasa (PE Applied Biosystems, Foster City, CA). Luego de la desnaturalización inicial (94°C-4 min) se realizaron 35 ciclos (desnaturalización: 94°C-60 seg/ pegado de primers: 55°C-60 seg/ elongación: 72°C-30 seg) y una extensión final de 72°C-10 min en un termociclador MJ Research (MJ Research, Watertown, MA). Los productos de la amplificación fueron migrados en geles de agarosa al 4% (Nu Sieve, FMC, Rockland, ME), teñidos con bromuro de etidio y fotografiados con sistema “Polaroid”.
Resultados y Discusión:
La hembra mutante donde apareció originalmente la mutación fue cruzada con un macho B6 para generar ratones híbridos F1 (D2B6F1), paso inicial del esquema de retrocruzas con el que fue creada la línea congénica (Figura 1)
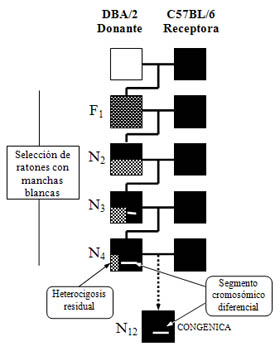
Figura 1: Esquema de cruzamientos utilizado para la construcción de la cepa congénica B6.D2-KitW/+ a lo largo de seis años de retrocruzas con selección del fenotipo de manchas blancas. A partir de la generación número 10 de retrocruzas se considera a la cepa como totalmente congénica, es decir que el segmento introducido (en promedio 20 cM) de la cepa dadora se encuentra ahora en un fondo genético con más de 99% de pureza de la cepa receptora (C57BL/6).
Los porcentajes de ratones “manchados vs. salvajes” obtenidos en estas cruzas no se alejaron nunca del 50% esperado, lo que permitió confirmar el carácter de mutación dominante (Figura 2).
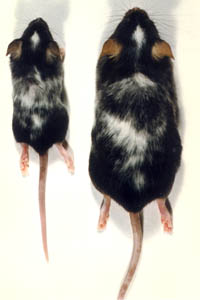
Figura 2: La fotografía muestra ratones B6.D2-KitW/+ (N12) de 30 días (izquierda) y 4 meses (derecha) de edad en posición dorsal. El fondo genético de estos ratones es >99% C57BL/6. Observar las manchas de pelaje blanco en la cabeza y dorso características de esta mutación.
El análisis de segregación del marcador microsatélite D5Nds2, ubicado a 41.0 cM del centrómero (cromosoma 5), en los ratones N13 sugiere que la mutación aparecida espontáneamente en nuestro bioterio era la mutación autosómica W, localizada en el mismo intervalo del cromosma 5 (75.8 Mb; 42.0 cM). De las 30 muestras de ADN de ratones con fenotipo de manchas blancas (supuestos KitW/+), 28 presentaron un genotipo heterocigota (2 bandas) 166/176 pb para el marcador D5Nds2. Esto demostró un ligamiento entre el alelo de 176 pb (de origen DBA/2) y la mutación en estudio, aportada en este caso por la cepa DBA/2 (Figura 3). Este fenómeno se explica porque el segmento del cromosoma original (DBA) que porta la mutación se mantiene intacto (ligamiento genético) a lo largo del desarrollo de la línea congénica. Las dos muestras restantes con genotipo homocigota 166/166 pb (banda única) fueron consideradas recombinantes. Las 30 muestras de ADN provenientes de ratones con un fenotipo normal –es decir sin manchas- (+/+) presentaron el genotipo esperado homocigota para el alelo B6 (banda única de 166 pb) para el marcador D5Nds2. No fue posible obtener ratones KitW/KitW por lo que se la consideró letal (no-viable) en estado homocigota.
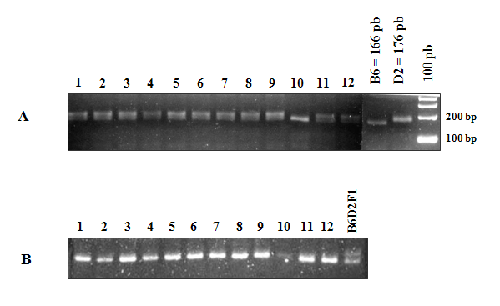
Figura 3: Geles de agarosa al 4% donde se observan los productos de amplificación por PCR del microsatélite D5Nds2. Este marcador molecular es polimórfico entre las cepas parentales [alelo B6 = 166 pb; alelo D2 = 176 pb). A. Las muestras 1-12 corresponden a ADN de ratones de fenotipo mutante (KitW/+). Notar que todas las muestras son heterocigotas 166/176 pb, excepto la muestra número 10, (homocigota 166/166) que es un recombinante. B. Las muestras 1-12 corresponden a ADN de ratones de fenotipo salvaje –sin manchas- (+/+), todos son homocigotas 166/166 pb. A la derecha, ADN de un ratón híbrido F1 (B6D2F1).
El piebaldismo (“piebald trait”) es una enfermedad hereditaria humana que presenta una deficiencia en la migración de los melanocitos hacia la piel y el oído interno originando defectos en la pigmentación del pelo e hipopigmentación en la piel de la frente, pecho y abdomen. Numerosas mutaciones en el oncogén Kit son las responsables del piebaldismo en los humanos y del Dominant White Spotting en el ratón. El locus Kit se localiza en el cromosoma 5 del genoma murino y se han identificado alrededor de 90 alelos diferentes. Los ratones heterocigotas exhiben manchas blancas en la cabeza y el vientre, un fenotipo muy similar al del piebaldismo humano. Los ratones homocigotas (KitW/KitW), en su inmensa mayoría, mueren durante el desarrollo embrionario. Sólo algunos alelos, denominados viables (v), permiten la sobrevida de los ratones homocigotas KitW-v/KitW-v, aunque éstos son anémicos, estériles, sordos y de pelaje blanco, pero, a diferencia de los albinos, tienen ojos oscuros, ya que no está afectada la migración de los melanocitos hacia la retina. Esta mutación tiene aplicaciones como modelo para el piebaldismo hereditario y en estudios experimentales de anemia (5).
Literatura Citada:
-
Benavides F. y Guénet J-L, Editors. Manual de Genética de Roedores de Laboratorio: Principios Básicos y Aplicaciones. Laboratory Animals Ltd., London, U.K., SECAL and Universidad de Alcalá, España, 2003.
-
Guénet J-L. Chemical mutagenesis of the mouse genome: an overview. Genetica 122: 9-24, 2004.
-
Benavides F., Stern M., Glasscock E., DiGiovanni J., Coghlan L., Conti C. Microsatellite DNA variants between the inbred SENCAR mouse strains. Molecular Carcinogenesis 28: 191-195, 2000.
-
Silver L. M. (ed.) Mouse Genetics. Concepts and applications. Oxford University Press, Oxford, 1995. (Versión online disponible en http://www.informatics.jax.org/silver/)
-
Fleischman RA. From white spots to stem cells: the role of the Kit receptor in mammalian development. Trends Genet 9: 285-90, 1993.


















Comentarios
Hola, muy buen artículo, ahora entiendo porque este tipo de raza duerme tanto, me gustaría preguntar si es que estos ratones pueden transmitir o ser infectados por el virus hanta, quedó atenta a sus comentarios, gracias!